Monitoreo de la elevación de las variantes del SARS-CoV-2 en las aguas residuales mediante la secuenciación del genoma
En un estudio reciente publicado en medRxiv * Servidor de preimpresión Los investigadores están realizando un seguimiento del genoma de las aguas residuales para detectar el síndrome respiratorio agudo severo coronavirus 2 (SARS-CoV-2) después de la aparición de las variantes alfa y delta en el Reino Unido.
estancia: Monitoreo genético de aguas residuales en toda la ciudad mediante la aparición secuencial de variantes alfa y delta del SARS-CoV-2. Haber de imagen: Aleksandar Malivuk/Shutterstock.com
Introducción
La infección por SARS-CoV-2, responsable de la enfermedad por coronavirus 2019 (COVID-19), se detectó principalmente mediante el ensayo de reacción en cadena de la polimerasa con transcriptasa inversa (RT-PCR). Sin embargo, al principio de la pandemia de COVID-19, las pruebas de SARS-CoV-2 entre personas infectadas y portadores asintomáticos fueron un desafío.
La mayoría de estos desafíos fueron causados por la pequeña cantidad de centros de prueba, la falta de personal capacitado para realizar la prueba, la falta de coordinación y gestión, la vacilación, la falta de incentivos entre las personas para realizar la prueba, así como la falta de consumibles para la prueba. pruebas Cada uno de estos desafíos se vio amplificado por la falta de recursos disponibles durante los primeros cierres.
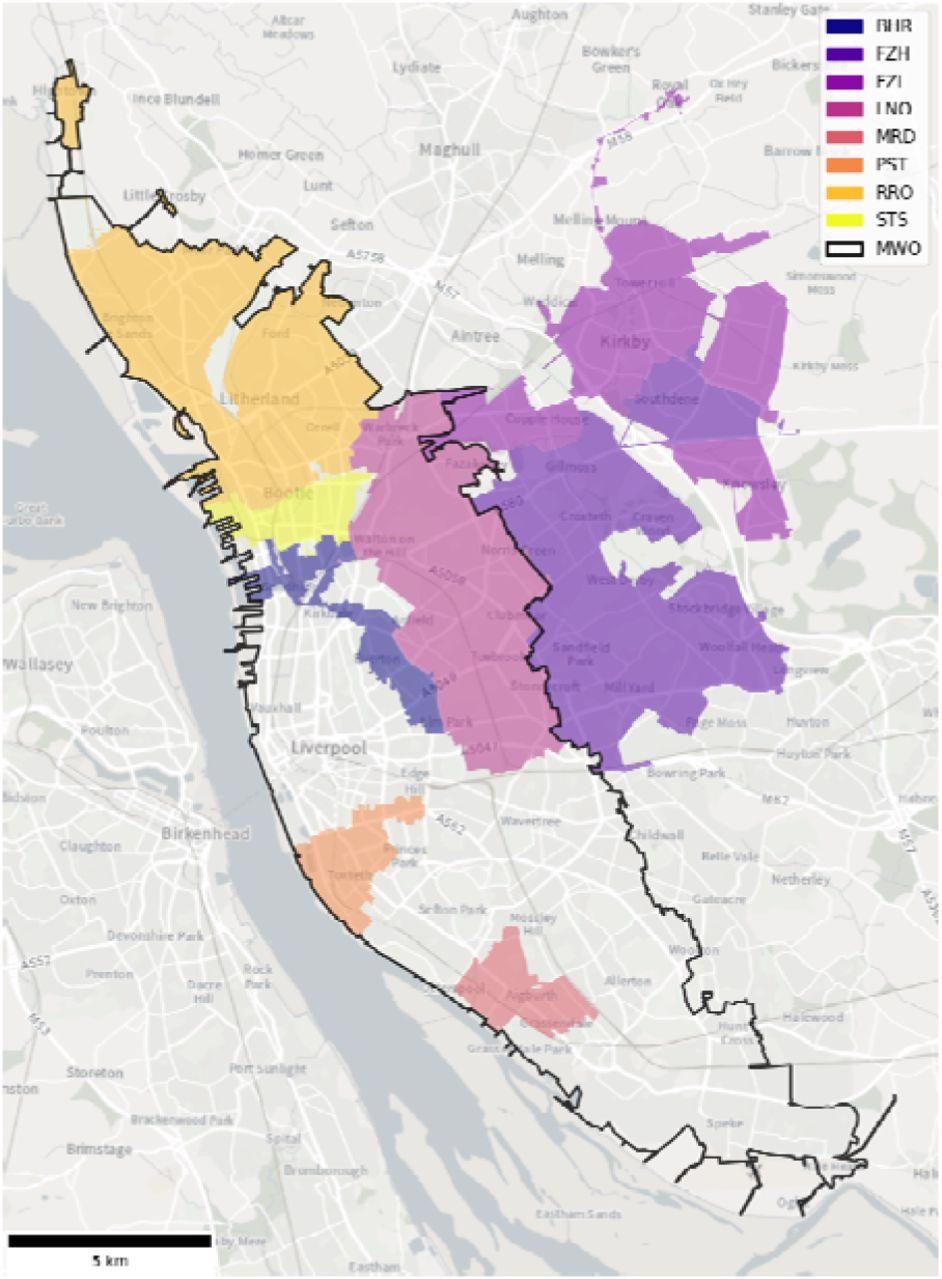
Captaciones de aguas residuales en 8 sitios de muestreo de aguas residuales (en color) y una planta de tratamiento de aguas residuales (MWO, contorno negro) en Liverpool. BHR = Bank Hall Relief, FZH = Fazakerley High, FZL = Fazakerley Low, LNO = Liverpool North, MRD = Mersey Road, PST = Park Street, RRO = Rimrose, STS = Strand SSO, MWO = Sandon Dock Main Works.
El papel de la monitorización genómica
Recientemente, el monitoreo genético se ha convertido en una herramienta útil para detectar el SARS-CoV-2 y sus variantes. El seguimiento genético consiste en secuenciar el material genético del agente infeccioso para identificar cualquier nueva mutación que pueda dar lugar a una nueva variante viral. Antes de la pandemia de COVID-19, el monitoreo genético se usaba principalmente para detectar microorganismos como las bacterias resistentes a los antibióticos y el virus de la influenza A.
Los datos de casos clínicos que utilizan la vigilancia genómica ayudan en las decisiones de salud pública al proporcionar información sobre la transmisión del virus, particularmente durante la aparición de nuevas variantes. Hasta la fecha, algunas de las variantes de SARS-CoV-2 más conocidas descubiertas hasta la fecha incluyen B.1.1.7 (Alpha), B.1.351 (Beta), P.1 (Gamma) y B.1617.2 (Delta) y, más recientemente, la variante B.1.1.529 (Omicron).
Aunque las vacunas contra la COVD-19 han seguido siendo eficaces contra estas variantes, muchas de estas nuevas cepas del SARS-CoV-2 están asociadas con una mayor transmisibilidad, una enfermedad más grave, una disminución de los anticuerpos neutralizantes, una disminución de la respuesta terapéutica y/o una disminución de la eficacia de la vacuna. Por lo tanto, la vigilancia genética se vuelve esencial debido a su capacidad para detectar, identificar y rastrear variantes del SARS-CoV-2 para ayudar a los epidemiólogos a comprender y predecir este virus y ayudar a controlar su propagación.
La vigilancia genómica del SARS-CoV-2 se logra mediante la secuenciación del genoma completo (WGS) de aislados clínicos utilizando ARN residual de RT-PCR de diagnóstico. Se han secuenciado y publicado más de siete millones de genomas de SARS-CoV-2 hasta enero de 2022 en la base de datos GISAID. La disponibilidad de esta secuencia del genoma ha proporcionado información sobre la evolución y la epidemiología del SARS-CoV-2.
Ventajas de la vigilancia basada en aguas residuales
A pesar de estas ventajas, la secuenciación clínica es muy costosa, incluso en países desarrollados. Por lo tanto, el monitoreo basado en aguas residuales como un enfoque complementario y rentable para la secuenciación clínica se ha utilizado cada vez más durante la pandemia de COVID-19.
Más del 50 % del SARS-CoV-2 se excreta en las heces; Por lo tanto, el ARN del SARS-CoV-2 se puede extraer y cuantificar mediante RT-qPCR mediante muestreo en plantas de tratamiento de aguas residuales o dentro de los sistemas de alcantarillado. Los resultados de este tipo de pruebas son consistentes con el auge y la caída de los casos clínicos.
En un estudio reciente publicado en medRxiv * Servidor de preimpresión, los investigadores demuestran que el monitoreo genético de las aguas residuales se puede usar de manera confiable para detectar la aparición de las variantes alfa (B.1.1.7) y delta (B.1.617.2) del SARS-CoV-2.
sobre estudiar
Se recolectaron muestras de aguas residuales de ocho sitios en la ciudad de Liverpool en el Reino Unido entre noviembre de 2020 y junio de 2021, así como de cuatro plantas de tratamiento de aguas residuales en el sureste de Inglaterra entre septiembre de 2020 y enero de 2021 como control.
Se extrajo el ARN de estas muestras y se secuenció para el análisis bioinformático. Los polimorfismos de un solo nucleótido (SNP) y las inserciones/eliminaciones dentro de estas muestras se identificaron y filtraron contra las mutaciones características de las variantes del SARS-CoV-2.
A nivel de amplicón, los investigadores buscaron mutaciones de firma simultáneas, lo que mejora en gran medida la confianza en la detección de variantes, especialmente en frecuencias más bajas. Sin mutaciones únicas acompañadas junto con una variante particular, no se puede identificar una variante confiable.
Resultados
La frecuencia de los SNP/indels distintivos alfa (B.1.1.7) aumentó entre el 2 de noviembre de 2020 y el 28 de febrero de 2021. Esta observación es estrechamente consistente con el aumento de casos clínicos de infección por SARS-CoV-2 Alfa en Liverpool y sureste de Inglaterra. .
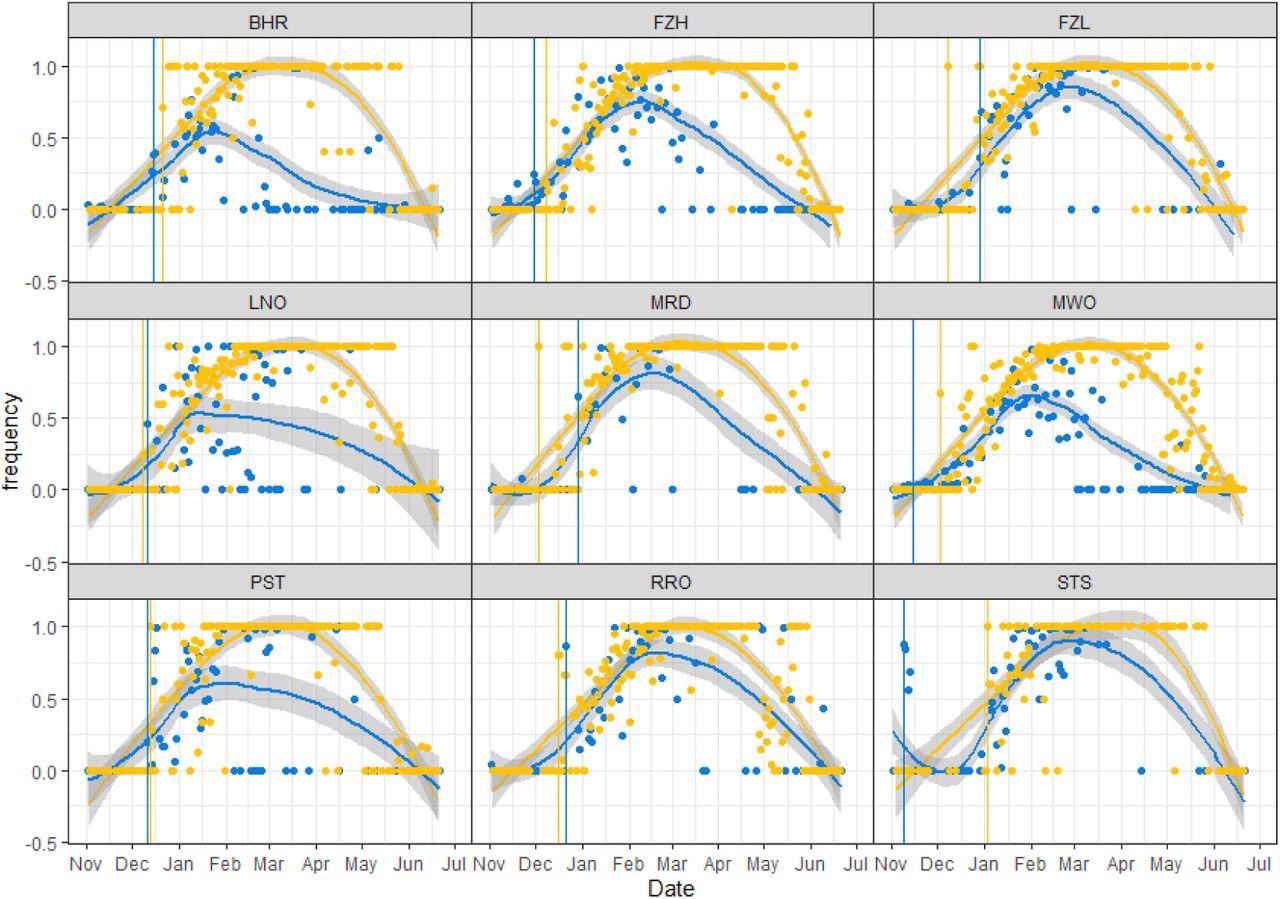
Frecuencia promedio de B.1.1.7 (Alpha) SNP/Indels detectados en aguas residuales versus muestras clínicas en cada cuenca, del 2 de noviembre de 2020 al 26 de junio de 2021. Los puntos muestran la frecuencia promedio de mutaciones alfa únicas para una muestra de agua residual determinada. azul) y la frecuencia de casos clínicos alfa a partir de una fecha determinada (amarillo). Las líneas de colores muestran el ajuste de la regresión polinomial local, incluidos los intervalos de confianza del 95 % sombreados. Las líneas verticales indican el primer caso clínico confirmado de Alfa (amarillo) y la primera detección en aguas residuales de mutaciones alfa concurrentes en el amplicón 147 (azul).
En la mayoría de los lugares, las frecuencias mensuales de aparición de alfa alcanzaron su punto máximo a mediados de diciembre, finales de enero y principios de febrero. Es importante destacar que la detección de aguas residuales alfa precedió a la detección clínica en el 56 % de los sitios de Liverpool hasta por 55 días, mientras que la detección clínica fue tan temprana como 26 días en los sitios del sudeste de Inglaterra.
A pesar de la detección temprana de variantes novedosas mediante el monitoreo de aguas residuales mediante análisis de redundancia, este enfoque es inconsistente. Si bien las razones de esto siguen sin estar claras, otros factores, incluidas las diferencias inherentes en los métodos de detección y las tasas de excreción de virus, así como la dilución debido a la precipitación, pueden contribuir a su inconsistencia.
A lo largo del mes se observan diferencias locales entre diferentes cuencas hidrográficas con frecuencias máximas temporalmente diferentes. Entre el 15 de marzo de 2021 y el 26 de junio de 2021, se observó un aumento significativo en las secuencias delta en todas las cuencas de captación de aguas residuales, con tendencias temporales variables en toda la ciudad. Este patrón coincidió con un aumento en la detección clínica de la infección delta y una disminución simultánea de la infección alfa.
Paralelamente a la disminución de infecciones en Liverpool desde abril hasta principios de junio de 2021, la detección de SARS-CoV-2 en muestras de aguas residuales también fue baja. Los investigadores también detectaron linajes esporádicos y bajos de SARS-CoV-2 B.1.351 (Beta), P.1 (Gamma), P.3 (Delta) y B.1.1.318 en el monitoreo de aguas residuales, que también son consistentes con la detección clínica de estas variables.
Conclusiones
Si bien la detección del monitoreo de aguas residuales presentó un patrón mixto de variables alfa en las regiones evaluadas, este enfoque de monitoreo tiene varias ventajas importantes, incluida la rentabilidad por unidad de población y un pequeño sesgo causado por la frecuencia de las pruebas. Sin embargo, la secuenciación genética de muestras clínicas ofrece mayor especificidad y potencial de rastreo de contactos.
En conjunto, el estudio actual destaca los beneficios de un enfoque complementario a la secuenciación genética mediante pruebas clínicas y monitoreo de aguas residuales. La detección temprana de nuevas variantes de SARS-CoV-2 a través de este método proporciona una dinámica precisa y fundamental de la cepa viral que se puede utilizar para medidas informadas de salud pública.
*Nota IMPORTANTE
medRxiv Publica informes científicos preliminares que no han sido revisados por pares y, por lo tanto, no deben considerarse concluyentes, orientar la práctica clínica o el comportamiento relacionado con la salud ni tratarse como información establecida.


