Los ingenieros están colocando miles de dispositivos de nanopartículas en una dirección precisa

Los investigadores han colocado más de 3.000 nanopartículas brillantes en forma de luna en un instrumento con forma de flor para indicar la polarización de la luz. Las «lunas» en cada uno de los doce pétalos apuntan en una dirección diferente y solo brillan cuando son golpeadas por luz polarizada que coincide con su dirección. El resultado final es una flor que ilumina secuencialmente sus pétalos a medida que la polarización gira de la luz que los ilumina. Venus, que es más pequeño que el ancho de un cabello humano, demuestra que miles de partículas pueden apuntar de manera confiable a la superficie de una oblea. Crédito: Ashwin Gopinath / Instituto de Tecnología de California
El proyecto de prueba de concepto allana el camino para la integración de partículas con chips de computadora.
Los ingenieros han desarrollado una tecnología que les permite colocar con precisión dispositivos microscópicos que consisten en un pliegue ADN Las partículas no solo están en una ubicación específica, sino también en una dirección específica.
Como prueba de concepto, han dispuesto más de 3.000 dispositivos moleculares a nanoescala brillantes en un instrumento con forma de flor para indicar la polarización de la luz. Cada uno de los 12 pétalos apuntaba en una dirección diferente alrededor del centro de la flor, y dentro de cada pétalo, aproximadamente 250 lunas estaban alineadas con la dirección del pétalo. Dado que cada luna solo se ilumina cuando choca con la luz polarizada que coincide con su dirección, el resultado final es una flor cuyos pétalos se iluminan en secuencia mientras rotan la polarización de la luz que los ilumina. Venus, que es más pequeño que el ancho de un cabello humano, demuestra que miles de partículas pueden apuntar de manera confiable a la superficie de una oblea.
Este método de posicionar y dirigir con precisión dispositivos moleculares basados en ADN puede hacer posible el uso de estos dispositivos moleculares para alimentar nuevos tipos de chips que integran biosensores moleculares con óptica y electrónica para aplicaciones como secuenciación de ADN o medición de concentraciones de miles de proteínas en tan solo uno.
La investigación, publicada por la revista el 19 de febrero de 2021 Ciencias, Se basa en más de 15 años de trabajo de Paul Rothemund del Instituto de Tecnología de California (BS ’94), profesor de investigación en bioingeniería, ciencias de la computación, matemáticas, computación y sistemas neuronales, y colegas. En 2006, Rothemund demostró que se puede dirigir el ADN para que se pliegue en formas delicadas mediante una técnica llamada Origami de ADN. En 2009, Rothemund y sus colegas de IBM Research Almaden describieron una técnica mediante la cual el ADN de origami podría colocarse en lugares específicos de las superficies. Para ello, utilizaron un proceso de impresión basado en haces de electrones y crearon parches «pegajosos» del mismo tamaño y forma que el origami. En particular, mostraron que los triángulos de origami están adheridos con precisión al sitio de los parches adhesivos triangulares.
A continuación, Rothemund y Ashwin Gopinath, anteriormente fue investigador postdoctoral senior en el Instituto de Tecnología de California y profesor adjunto en Con, Refinamiento y expansión de esta tecnología para demostrar que los dispositivos moleculares hechos con DNA Origami pueden integrarse de manera confiable en dispositivos ópticos más grandes. «La barrera tecnológica fue cómo se organizan repetidamente cantidades masivas de dispositivos moleculares en los patrones correctos en los tipos de materiales utilizados en los chips», dice Rothmond.
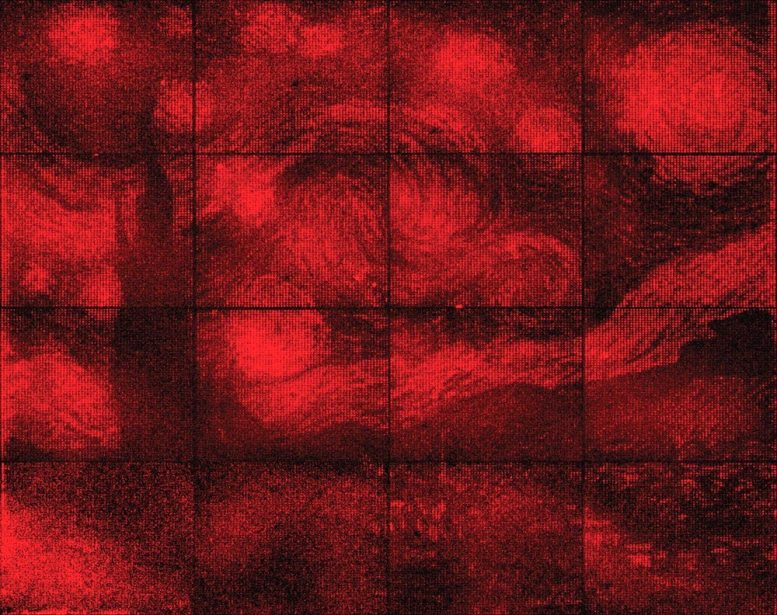
Esta brillante reproducción de «La noche estrellada» tiene 65.536 píxeles y un ancho de diez centavos la docena. Crédito: Ashwin Gopinath / Instituto de Tecnología de California
En 2016, Rothemund y Gopinath demostraron que se pueden usar partículas fluorescentes con triple origami para la reproducción. Versión de 65.000 píxeles de Vincent Van Gogh Noche estrellada. En este trabajo, se utilizó origami de triple ADN para colocar partículas fluorescentes dentro de resonadores de luz del tamaño de una bacteria. La posición exacta de las partículas fluorescentes era fundamental porque moverse solo 100 nm hacia la izquierda o hacia la derecha atenuaría o iluminaría el píxel más de cinco veces.
Pero la técnica tenía un talón de Aquiles: “Dado que los triángulos eran equiláteros y podían girar y voltearse libremente, podían adherirse planos al parche triangular adhesivo en la superficie de cualquiera de las seis formas diferentes. Esto significa que no podemos usar cualquier dispositivo que requiera orientación. Ponerse a trabajar. «Nos quedamos atrapados con dispositivos que funcionan igual de bien cuando se apuntan hacia arriba, hacia abajo o en cualquier dirección». Los dispositivos moleculares para la secuenciación de ADN o la medición de proteínas deben aterrizar en el lado derecho, por lo que Las viejas tecnologías del equipo arruinarían el 50 por ciento de los dispositivos. Para los dispositivos que también requieren una dirección de rotación única, como los transistores, solo el 16 por ciento funcionará.
El primer problema a resolver, entonces, fue hacer que el ADN Origami aterrizara de manera confiable con el lado derecho hacia arriba. «Es un poco como asegurarse de que la tostada siempre deje caer la mantequilla por arte de magia en el suelo», dice Rothmond. Para sorpresa de los investigadores, cubrir la alfombra de origami con una alfombra de hebras elásticas de ADN en un lado permitió que más del 95 por ciento aterrizara en su cara. Pero el problema con el control de la rotación permaneció. Los triángulos basados en tres longitudes diferentes fueron el primer intento de los investigadores en una forma que podría aterrizar en una rotación preferida.
Sin embargo, después de luchar para obtener solo el 40 por ciento de los triángulos rectángulos para indicar la dirección correcta, Gopinath reclutó a los informáticos Chris Thatchuk de Universidad de Washington, Coautor de Ciencias Paper y ex médico postdoctoral en el Instituto de Tecnología de California; David Kirkpatrick de la Universidad de Columbia Británica también es coautor de un libro Ciencias papel. Su misión era encontrar una forma que solo se mantuviera en la dirección prevista, independientemente de la dirección en la que aterrizara. La solución de los informáticos fue un disco con un orificio descentrado, que los investigadores llamaron «la pequeña luna». La evidencia matemática sugirió que, a diferencia del triángulo rectángulo, las lunas pequeñas pueden rotar suavemente para encontrar la mejor alineación con su parche adhesivo sin quedarse atascado. Los experimentos de laboratorio demuestran que más del 98 por ciento de los satélites pequeños encuentran la orientación correcta para sus puntos adhesivos.
Luego, el equipo agregó partículas fluorescentes especiales que se apiñan firmemente en las espirales de ADN de las lunas pequeñas, perpendiculares al eje de las espirales. Esto asegura que todas las partículas fluorescentes dentro de la luna apunten en la misma dirección y brillarán más intensamente cuando sean estimuladas por una luz polarizante específica. “Es como si cada molécula que lleva una pequeña antena pudiera aceptar la energía de la luz de manera más eficiente solo cuando la polarización de la luz coincide con la dirección de la antena”, dice Gopinath. Fue este simple efecto el que permitió la construcción de la flor sensible a la polarización.
Con métodos robustos para controlar la dirección ascendente y de rotación del origami de ADN, ahora se puede integrar una amplia gama de dispositivos moleculares a bajo costo en chips de computadora de alto rendimiento para una variedad de aplicaciones potenciales. Por ejemplo, Rothemund y Gopinath fundaron una empresa, Palamedrix, para comercializar la tecnología para construir chips semiconductores que permitan el estudio simultáneo de todas las proteínas relevantes para la salud humana. El Instituto de Tecnología de California presentó solicitudes de patente para el trabajo.
Referencia: «La orientación absoluta y arbitraria de las formas de una sola molécula» por Ashwin Gopinath, Chris Thatchuk, Anya Mitskovets, Harry A. Water, David Kirkpatrick y Paul W.K Rotemund, 19 de febrero de 2021, Ciencias.
DOI: 10.1126 / science.abd6179
Autores del Instituto de Tecnología de California: 20181029-101527551
El título del artículo es «Dirección absoluta y arbitraria de las formas monomoleculares». Los coautores de Caltech incluyen a Harry Atwater, el profesor de física aplicada y ciencia de los materiales Howard Hughes, y la ex estudiante de posgrado Anna Mitskovitz (Ph.D.20). Este trabajo fue apoyado por la Oficina de Investigación Naval, la Oficina de Investigación Científica de la Fuerza Aérea, la Fundación Nacional de Ciencias, la Fundación Or Family, el Instituto Abdeen y la Beca Postdoctoral Banting.

