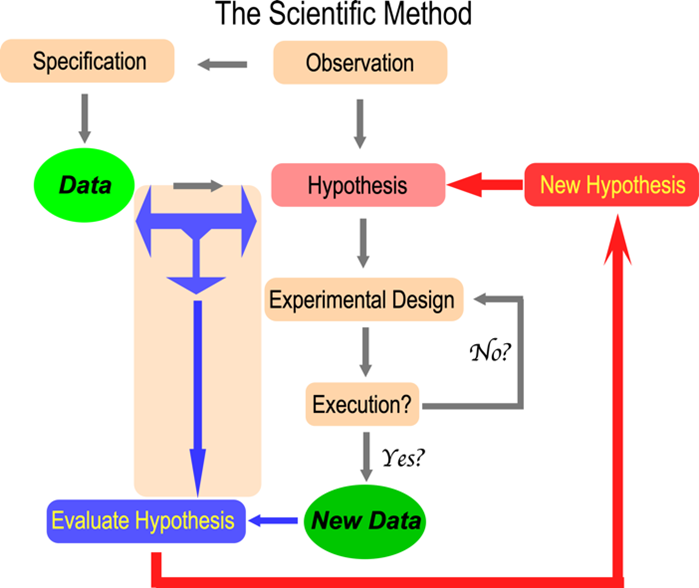
Explorando la dinámica del método científico en la codificación genética
El profesor Charlie Carter del Departamento de Bioquímica y Biofísica de la Universidad de Carolina del Norte en Chapel Hill está buscando la forma científica de comprender el origen de la codificación genética, y más
profesor charles carter Se formó en cristalografía molecular de rayos X con Joseph Kraut en UCSD y con Aaron Kluge en el Laboratorio de Biología Molecular del Consejo de Investigación Médica en Cambridge, Reino Unido.
Como sugiere la imagen, la dinámica del método científico fascina al profesor Carter. su investigacion Ha contribuido ampliamente a la biología estructural, el diseño experimental, las mecanoenzimas (acoplamiento activo dentro de las proteínas), la bioinformática y la biología evolutiva, el origen de la codificación genética.
Diseño experimental en investigación.
La mayoría de los procesos de interés experimental dependen de múltiples factores. Varios factores a menudo interactúan. Estas interacciones son difíciles de resolver. Mi padre me presentó un diseño experimental factorial al examinar el crecimiento de cristales. Me enseñó a reducir los alias para los diseños de factores parciales. Nuestro artículo (1) inspiró todos los exámenes posteriores del crecimiento de cristales. Sigue siendo mi artículo más citado.
El cambio de fase (2) limitó el valor de muestreo al producir fases de alta precisión usando solo el 1% de las combinaciones posibles. Neil Sloan (Bell Labs) me introdujo a los experimentos de respuesta de superficie, permitiéndonos explorar la vecindad de los óptimos (3,4). Un trabajo reciente identificó un aspecto inesperado de la edición del ARNm por la citidina desaminasa APOBEC1: el agente proteico redujo la temperatura óptima de 42 °C a 37 °C, ¡que es la temperatura a la que viven los humanos!
Aminoacil-tRNA sintetasa (aaRS) y el origen de la catálisis y la codificación de genes
Los estudios de la biología estructural del complejo triptofanil-tRNA nos llevaron a estudiar cómo evolucionaron las enzimas que traducen el código genético (5,6). Utilizamos el software de diseño de proteínas Rosetta para diseñar los fragmentos más conservados de aaRS de clase I y II como catalizadores solubles.
Las estructuras resultantes contienen 130 residuos (7). Los llamamos orzimas. Acelera la activación de 10 aminoácidos9Plegar y conservar las funciones de activación de aminoácidos y acilación de ARNt necesarias para la traducción simple de codones (8).
Las proteozimas (46 residuos) son estructuras más pequeñas anidadas dentro de las orzimas. Acelera la activación de 10 aminoácidos6– doblar (9). La eficiencia catalítica de las dos clases de aaRS aumenta linealmente con la longitud de la secuencia (9). Estas optimizaciones paralelas son clave para sincronizar diferentes reacciones químicas en las células con un mayor desarrollo de enzimas.
Varias mediciones biofísicas indican que las orzimas son glóbulos fundidos catalíticamente activos (10). Por lo tanto, era probable que los primeros biocatalizadores fueran polipéptidos parcialmente plegados y flexibles que pueden formar transitoriamente enlaces estrechos para estados de transición de reacciones químicas.
Codificación génica bidireccional para aminoacil-tRNA de clase I y II
Confirmamos tres predicciones de la extraña hipótesis (11) de que los genes ancestrales de aaRS de clase I y II eran hebras opuestas del mismo gen. (i) Las partes más conservadas (es decir, las urzimas) son exactamente las que corresponden a la codificación bidireccional.
Su fuerte actividad motivacional es necesaria para que la hipótesis sea válida (5). (2) Quedan rastros de codificación bidireccional en los genes aaRS contemporáneos. Esta señal aumenta a medida que las reconstrucciones independientes de las dos superfamilias se acercan al nodo raíz (12). (3) Los 46 péptidos restantes que contienen sitios de unión a ATP de aaRS clase I y II, expresados a partir de un gen bidireccional de diseñador, tienen las actividades catalíticas esperadas (9).
Además, los péptidos de 46 residuos tienen una actividad catalítica significativa. Los péptidos del gen de ingeniería bidireccional lograron la misma eficiencia catalítica con kgato DE ACUERDOMETRO Valores 100 veces superiores a los encontrados en secuencias WT de tipo salvaje extraídas de las estructuras originales de Clase I TrpRS y Clase II HisRS. Los cuatro péptidos aceleran la activación de aminoácidos por ATP en la misma cantidad. Por lo tanto, este experimento es una prueba directa exitosa de la hipótesis de codificación de significado/antisentido de Rodin y Ohno.
Acoplamiento alostérico y transferencia de energía libre.
El movimiento de dominio impulsa la activación de aminoácidos a través de la síntesis de triptofanil-tRNA. La removilización de la cadena lateral aromática en una “clave principal” (clave D1) media las fuerzas de cizallamiento inducidas por el movimiento del dominio. La recarga limita la tarifa. También impone un comportamiento multiestado durante la estimulación. La removilización, a su vez, cambia la orientación del dominio al sitio activo, activando el sitio activo de Mg2+ Ión (13).
El movimiento del dominio configura transitoriamente el sitio activo para completar el estado de transición de la reacción. Los ciclos termodinámicos estándar y la mutación conformacional miden las energías libres de acoplamiento interno que impulsan tanto la catálisis (13–15) como la especificidad (16). Los movimientos de dominio mejoran tanto la motivación como la especificidad (16) en la misma medida que el acoplamiento entre las cadenas laterales de los interruptores principales D1 individuales y el sitio activo (13, 14). La dependencia conformacional general de ΔG en la liberación de PPi se combina con la integración del estado de transición para una utilización eficiente de ATP.
El ‘mecanismo de equilibrio’ resultante es un caso especial de activación recíproca, que es un fenómeno más general, que asegura la sublimidad vectorial de la segunda ley de la termodinámica (17). Unifica los orígenes de la catálisis, la genética y la bioenergética (18).

“Defensor de la Web. Geek de la comida galardonado. Incapaz de escribir con guantes de boxeo puestos. Apasionado jugador”.